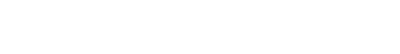La industria del biochip
La introducción de material biológico en un chip ha dado lugar a una floreciente industria bioinformática con aplicaciones en la medicina, inteligencia artificial y computación, entre otras disciplinas
Evolución histórica
El biochip se fundamenta en la inmovilización de material biológico, de ahí el término bio, sobre una superficie sólida para realizar un ensayo de afinidad entre el material inmovilizado sonda y el material de muestra blanco.
Todo lo que engloba la palabra biochip tuvo su inicio en la década de 1960, al inmovilizar muestras genéticas sobre soportes sólidos. Una década más tarde, Edwin Southern utiliza como soporte sólido filtros de nitrocelulosa, sobre los que se adherían las moléculas de ADN (ácido desoxiribonucleico). Se eliminaba de esta forma el problema que se tenía de interacción del ADN con otras moléculas inmovilizadas, pero permanecía la hibridación con las otras moléculas complementarias de disolución. En cuanto a la detección de los puntos en los que se producía la hibridación, se hacía mediante un marcador radiactivo en un revelado por autorradiografía. Esta técnica es conocida como Southern Blot. Si seguimos avanzando en el tiempo hacia la aparición del biochip, el siguiente paso consistió en la construcción de matrices de material biológico inmovilizado con la técnica Soutern. Ya se comienza a hablar de estructuras matriciales y se hace necesaria la utilización de materiales porosos, entre los que se encuentran las membranas de nitrocelulosa o nylon. Sobre esos poros o puntos de la matriz, se depositará posteriormente el material biológico a inmovilizar. A partir de ahora se puede decir que aparece el término conocido como densidad o nivel de integración, para darnos una idea de los tamaños de los poros y de su proximidad. Lógicamente, a partir de entonces las condiciones de diseño van a estar orientadas a un tamaño de los poros lo más pequeño posible, al igual que la distancia existente entre ellos. No olvidemos, que las tecnologías electrónicas actuales están tocando techo y que no se puede conseguir mucho más de lo que existe actualmente, en cuanto a nivel de integración y velocidad de procesamiento.
Por otro lado, aparecen soportes como el vidrio y el silicio y comienza el desarrollo de las micromatrices.
A finales de la década de 1980 aparece la técnica Genechip, desarrollada por Affymetrix, con la que se alcanza una gran capacidad de miniaturización. Su inicio tuvo lugar en la compañía Affymax por un grupo de científicos: Stephen Fodor, Michel Pirrung, Leighton Read y Lubert Stryer, que trabajaban en la síntesis sobre superficies sólidas de péptidos. En la actualidad se han desarrollado nuevas técnicas o se han perfeccionado las que existían anteriormente, lo que ha permitido alcanzar el alto grado de diversidad tecnológica existente hoy en día, dando lugar a la aparición de numerosas compañías en el sector (tabla 1).
En nuestros días, el término biochip está siendo utilizado en una gran variedad de campos científicos, debido a su diversidad de aplicaciones, que han dado lugar a la aparición de términos como la biocomputación y la bioinformática.
Ejemplos de aplicación de estas nuevas tecnologías son: las unidades de memoria basadas en proteínas, los procesadores neuronales y la computación basada en ADN.
En cuanto a la formación del personal especializado, se hace cada vez más necesario técnicos formados en biología molecular, informática y electrónica, para poder desarrollar nuevos sistemas de información (algoritmos y software) y tecnologías de computación (hardware).
Bioinformática
La bioinformática, como su nombre indica, se encarga de aplicar las tecnologías informáticas para resolver problemas de carácter biológico. Es una interfase o puente entre la biología y la computación. Aunque inicialmente esta técnica “emergente” era sólo un mero soporte de almacenamiento y gestión de la gran cantidad de datos y conocimientos desarrollados en los diversos experimentos biológicos. En la actualidad, ya se están realizando desarrollos y simulaciones de experimentos en ordenadores. Su objetivo está centrado en trabajar en la investigación y desarrollo de herramientas útiles, que ayuden a comprender el flujo de información biológica originado en las estructuras moleculares, los genes, la conducta biológica, etc., para una posterior aplicación en campos como la biocomputación o los biodispositivos, englobando en estos últimos, aplicaciones como pueden ser entre otras, el control de los temblores en la enfermedad de Parkinson, los implantes cocleares, etc.
Centrándonos en las etapas de fabricación de un biochip, existen los llamados programas de clone tracking, encargados de guardar toda la información acerca de la localización de las sondas, su origen, etc. A continuación del revelado viene el almacenamiento de datos obtenidos, para utilizar en un posterior análisis y de esta forma conseguir una mejora del modelo a analizar. La mayoría de programas que se encargan del análisis y tratamiento de resultados emplean técnicas de minería de datos (data mining). La gran diferencia existente entre esta técnica y los programas de análisis se debe a que la minería de datos posee herramientas para la representación gráfica de resultados y modelos de análisis. Esta técnica puede extraer datos ocultos entre grandes cantidades de información, como pueden ser los patrones de comportamiento. Una de las técnicas de minería de datos más utilizada es el análisis por clustering, en la que se agrupan los datos según unos coeficientes de similitud como pueden ser diferentes algoritmos, coseno, etc.
En cuanto a los datos procedentes del revelado, se pueden presentar de forma numérica o en imagen de 16 bits (formato TIFF), apreciándose los puntos en los que la hibridación ha sido positiva y en los que no ha habido tal hibridación. En estas tres fases, que se pueden englobar dentro de una única, y estando todas ellas relacionadas con los datos obtenidos en el revelado, es donde se aplican con mayor profundidad los elementos de software bioinformático.
Para el diseño y análisis de resultados, provenientes de los experimentos con biochips, se han desarrollado un gran número de herramientas software a causa de la gran cantidad de información generada, repercutiendo en una mayor necesidad de que los programas sean cada vez más potentes y evolucionen hacia una mejora de resultados de forma permanente.
Por último, hay que decir que existen programas de tratamiento de imágenes que permiten analizar las entradas de varios chips simultáneamente.
Diseño biochip
El proceso de diseño o fabricación de un biochip se puede ver resumido en la figura 1.
Existen una serie de etapas en el proceso de fabricación, que han sido descritas la mayoría de ellas en el apartado de bioinformática. A continuación, se completará la descripción de estas fases de diseño partiendo de los comentarios hechos anteriormente:
Diseño del biochip
Donde se seleccionará el tipo y cantidad de material biológico, que se va a inmovilizar sobre la superficie del chip. En esta fase de trabajo, se seleccionarán los parámetros internos, según el tipo de ensayo. En cuanto al número de sondas o densidad de integración, se va a ver limitada según el método de fabricación a emplear.
Fabricación
En esta etapa es muy importante a tener en cuenta, si el biochip es “comercial” o “personalizado”. En el caso de que el biochip sea personalizado, tanto el proceso de diseño como el de fabricación, es el investigador el encargado de llevarlos a cabo, mientras que en el primer caso son realizados por el fabricante. La principal diferencia existente entre ellos, es que el experimento se tiene que adaptar a las características que ofrece el dispositivo si es comercial y, si es personalizado, el diseño del biochip se centrará en su adaptación a las necesidades del experimento para el que es necesario. Esta característica ofrece una ventaja de los personalizados frente a los comerciales contrarrestada con la mayor densidad de integración que ofrecen los comerciales frente a los personalizados. En cuanto a los equipos encargados de la fabricación de los biochips están los arrayers o robots, capaces de depositar cantidades microscópicas de material biológico sobre la superficie del chip. Estos robots vienen provistos de un software, para de esta forma poder inmovilizar con la mayor precisión y exactitud posible las sondas, según unas coordenadas prefijadas por el usuario.
Preparación de la muestra
Esta etapa se divide en tres fases y en cada una de ellas se desarrolla un proceso que permita su empleo o utilización posterior:
– Extracción y purificación del material a analizar: ADN, ARN (ácido ribonucleico) o proteínas.
– Amplificación, permitiendo multiplicar pequeñas cantidades de ADN y ofreciendo la ventaja de que el segmento de ADN que sirve de molde, no requiere estar necesariamente en estado puro, sino que puede ser parte de mezclas complejas. La sensibilidad de esta técnica es muy alta, pero por el contrario presenta algunos inconvenientes, uno muy a tener en cuenta es la alta probabilidad de obtener falsos positivos por contaminación.
– Marcaje de la muestra, necesario para permitir la detección de los puntos donde se ha producido la hibridación en el proceso de revelado. Los marcadores más comúnmente empleados son los fluorescentes, pero también puede utilizarse marcaje radioactivo o quimioluminiscencia. Aunque es necesario el marcaje, su utilización impide el desarrollo de nuevas plataformas de biochips. Según los expertos, nuevos métodos de detección no basados en el marcaje de sondas, como pueden ser detección óptica, electroquímica o espectrometría de masas MALDI-TOF, serán determinantes a la hora de desarrollar nuevos biochips. Entre otras ventajas se conseguirían una mayor sensibilidad, reducción de las cantidades de muestras necesarias y, sobre todo, se podrían omitir los pasos previos de amplificación.
Hibridación y lavado
En estos dos procesos no existe gran diferencia en cuanto a utilización de biochip comercial o personalizado, exceptuando el tipo de tecnología a utilizar en uno u otro. La hibridación es clave, al tenerse que bus-car la máxima reacción de afinidad entre la sonda y su complementaria inmovilizada en la superficie del chip, para obtener los mejores resultados en el proceso de revelado. En cuanto al lavado, se realiza para eliminar las iteraciones inespecíficas, que se dan entre la muestra y el material inmovilizado o la superficie del biochip.
Revelado
Etapa donde existen numerosas alternativas y hay que tener claro la que va a proporcionar el mejor resultado. Entre las técnicas más comunes y económicas de revelado que se utilizan, están los isótopos radioactivos para el marcaje de los “blancos” y su posterior detección. En cuanto a la detección de marcadores fluorescentes con los que previamente se ha marcado la muestra, existen otras técnicas más sofisticadas, me estoy refiriendo a los escáneres láser y cámaras CCD para detectar los marcadores fluorescentes, alternativas ecológicas en contraposición a los marcadores radioactivos, pero con la desventaja de tener un mayor coste económico.
Técnicas y métodos de fabricación
Las técnicas de fabricación de los biochips se han diversificado mucho desde su origen, permitiendo establecer grandes diferencias entre los distintos dispositivos y plataformas existentes en los mercados.
Las matrices de material genético podemos clasificarlas según su densidad de integración en micromatrices y macromatrices. En las micromatrices la densidad es mucho más alta, debido a que las técnicas de generación de puntos en la matriz poseen un mayor avance tecnológico. En contrapartida, las macromatrices ofrecen una mayor sencillez de fabricación. Estas grandes diferencias en la capacidad de integración entre ambos dispositivos van a generar una “selección” de posibles aplicaciones de unas y de otras.
El soporte y tipo de unión sobre el que se va a generar la matriz es un componente muy importante, ya que según su resistencia, se podrán llevar a cabo determinados procesos de lavado y eliminación de uniones inespecíficas, determinándose también si van a ser reutilizables en procesos posteriores, la realización de un mecanismo de revelado u otro y un aspecto muy importante a tener en cuenta, el coste que conlleva la fabricación de la matriz. Si queremos hacer una clasificación teniendo en cuenta las características anteriores, los chips se pueden clasificar en tres grupos:
– Glass-based: la superficie sólida (cristal, silicio, plástico, oro) sirve de soporte y sobre ella se encuentra el material covalentemente inmovilizado.
– Gel-based: los soportes utilizados son membranas porosas de nylon o nitrocelulosa y pequeñas porciones de geles. Por consiguiente, la iteracción no tiene carácter covalente.
– Electronic-based: el soporte en este caso es un electrodo. La iteracción entre las moléculas a inmovilizar y los electrodos es de tipo electrostática.
En la etapa de inmovilización del material genético en la superficie y formación de la matriz, existen dos procesos claramente diferenciados por el modo en que se sintetiza el material genético de las sondas y se fabrican las matrices:
Sintetización externa
El material genético de las sondas es sintetizado como su nombre indica exter-namente a la matriz, para depositarlo posteriormente sobre la superficie. Este método ha impulsado el desarrollo de robots (arrayers) para la generación de las matrices y ha supuesto un aumento en la capacidad de producción de matrices, pero no es la única que emplea un material ya sintetizado.
Sintetización interna
El material genético es sintetizado in situ sobre el substrato. Consiste en la unión química de la muestra sobre la superficie de la matriz.
Ante la imposibilidad de describir todas las tecnologías de fabricación por las limitaciones en cuanto a extensión que ofrece la publicación, voy a centrarme en algunas de las diferentes alternativas que existen, seleccionando algunas de las tecnologías más relevantes en la actualidad para fabricar un biochip (tabla 2).
Fotolitografía
La fabricación de los biochips está inspirada en el método de fabricación de los chips electrónicos. En estos últimos, mediante la fotolitografía, se depositan circuitos microscópicos, y en los biochips lo que se depositan son cadenas de ADN.
El método consiste en la síntesis in situ de las cadenas a inmovilizar, generalmente sobre un soporte de vidrio o cristal. Estas cadenas generalmente están compuestas por ácidos nucleicos, que posteriormente se unirán al grupo de reactivos.
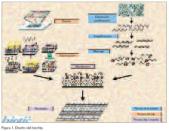
La utilización de la máscara y su forma hace posible que la luz incida sobre las posiciones y regiones donde sea necesaria la construcción de cadenas de material genético. Esta máscara previamente diseñada, será transparente para una determinada longitud de onda, y opaca en las zonas a proteger del proceso fotolitográfico (figura 2). A continuación hay que añadir al chip los cuatro nucleótidos: adenina, guanina, timina y citosina (A, G, T, C), que se unirán a los grupos reactivos contenidos en el cristal mediante enlace covalente. Hay que decir que este proceso se repetirá una y otra vez con sus correspondientes lavados, hasta generar los oligonucleótidos con las secuencias que requiera el proceso. En la figura 3 aparecen de forma gráfica y ordenada todos los pasos a realizar. En esta figura se puede ver que tras la hibridación se hace necesario eliminar los residuos o fragmentos que no han hibridado mediante un lavado.
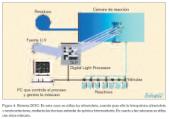
Existen también otros métodos fotolitográficos, que no emplean los patrones de máscara sino un sistema llamado DOC (Digital Optical Chemistry) (figura 4). Esta técnica ha sido desarrollada de manera conjunta por Southwestern University Medical Center y la empresa tecnológica Texas Instruments.
El chip utilizado por el sistema DOC contiene unos microespejos, cuyo número puede variar entre varios miles de centenares o millones, controlados digitalmente por un ordenador, de acuerdo con el patrón de secuencias que se desee preparar. Estos microespejos se encargan de localizar el haz de luz ultravioleta sobre el substrato de cristal, que va a actuar como soporte de la matriz.
En la fabricación por este procedimiento se utiliza el procesador digital de luz DLP (Digital Light Processor), un mecanismo de soporte de los substratos de las matrices y que además controla la dispensación de reactivos para la síntesis de los oligonucleótidos.
Robots piezoeléctricos
El principio de funcionamiento de este método es el mismo que el utilizado para la fabricación de las impresoras de inyección de tinta. En cuanto a los reactivos, son depositados en los puntos seleccionados mediante el movimiento de los cabezales, que contienen los inyectores, controlados por un robot. Estos inyectores piezoeléctricos o micropipetas piezoeléctricas, son capaces de producir la inyección de las sondas de material genético. Entre los parámetros claves a controlar en estos dispositivos: todos los inyectores tienen que depositar la misma cantidad de reactivo en todas las celdas de la matriz y que el brazo de robot tiene que desplazarse unas distancias muy cortas pero muy precisas (figura 5).
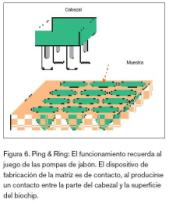
Ping & Ring
Este sistema utiliza un sencillo mecanismo, lo que permite que su costo sea bajo y además su montaje es fácil de realizar en arrayers de laboratorio. Esta técnica fue desarrollada y comercializada por la empresa Genetic Microsystems, para posteriormente ser absorbida por Affymetrix. Como se puede ver en la figura 6, el arrayers va provisto de una serie de cabezales, cuyas partes principales son un anillo o aro y una aguja, encargada de producir la impresión. El proceso de impresión de la muestra sobre la superficie comienza al introducir el anillo en la muestra, quedando impregnado de ésta debido a la tensión superficial del líquido. Parte de la muestra queda retenida en la parte hueca del anillo. A continuación, la aguja pasará a través del anillo, arrastrando el material retenido en el centro de éste, para posteriormente ser depositado sobre la superficie del biochip.
Al necesitarse que el material de la muestra esté previamente preparado, o lo que es lo mismo, que esté previamente sintetizado para su inmovilización, este mecanismo no permite la síntesis in situ.
Presente y futuro
Como es sabido por todos, las actuales tecnologías microelectrónicas están tocando techo en las máximas prestaciones que pueden dar en cuanto a velocidad y densidad de integración. Aunque existen graves inconvenientes como puede ser que la longitud de onda empleada en la fotolitografía limita el tamaño de los componentes que se pueden crear con este procedimiento. Para fabricar componentes electrónicos más pequeños, hasta una décima parte de una micra de diámetro (actualmente estamos sobre una quinta parte), es necesario utilizar una longitud de onda más corta, como puede ser luz ultravioleta de menor longitud, los rayos X o los haces de electrones de alta energía.
Otro obstáculo a eliminar es el grosor de la capa de dióxido de silicio que actual-mente está en torno a los 24 átomos de grosor, y se quiere llegar a conseguir como mucho que el grosor sea de 5 átomos. Con el inconveniente de que este óxido tan fino no es capaz de aislar las capas de silicio, tendría tantas pérdidas que sería inútil. Incluso aislantes con el doble de grosor, empezarían a romperse si se fabricasen con los métodos actuales.
En cuanto a la parte económica, la industria no puede cambiar sus cadenas de producción y sus inversiones rápidamente. Al exigirse un alto coste, actual-mente se tiende, como se ha hecho siempre, a una adaptación progresiva entre las tecnologías que existen actualmente y las que existirán en un futuro. Creándose un puente o interfaz, basado en tecnologías híbridas entre semiconductores, moléculas orgánicas y metales, permitiendo de este modo, que el salto hacia las tecnologías del futuro se haga de una forma menos traumática.
Aplicaciones
Aunque el campo de aplicaciones se puede decir que no tiene límites y a medida que se avanza en el estudio de estos dispositivos se abren nuevas expectativas, sirvan como ejemplo las siguientes:
– Computación: científicos israelitas han construido una computadora con ADN. Sus principales características a destacar son: un millón de ellas podría caber en un tubo de ensayo, realización de mil millones de operaciones por segundo con un 99,8 por ciento de precisión. Otra aplicación son las memorias moleculares en la que actualmente trabajan los laboratorios de IBM.
– Medicina: los implantes cocleares para corregir anomalías en el oído. El Oncochip, que va a permitir entre otras cosas saber si una persona está predispuesta genéticamente a padecer cáncer, o en qué fase de desarrollo se encuentra la enfermedad, para aplicar la medicación adecuada.
– Inteligencia artificial: los “algoritmos genéticos” en que la computadora simula el funcionamiento neural biológico. A destacar es también el trabajo que están realizando los ingenieros de la NASA en el diseño de computadoras del tamaño de una molécula de proteína, que servirán para rastrear y reparar daños celulares en el organismo humano.
Estandarización
Al existir una gran diversidad de recursos y de tecnologías disponibles, la consecuencia más inmediata ha sido una aparición de diferentes técnicas de fabricación, dimensiones, tecnologías posibles, etc., con la consiguiente incompatibilidad entre las diferentes plataformas. La solución está en una estandarización tanto a nivel de software como de hardware, que garantice una compatibilidad o comprensión entre las diferentes técnicas de fabricación.
En la actualidad destacan tres propuestas de estandarización:
– Primera iniciativa en 1998 por parte del consorcio GACT (Genetic Analisys Technology Consortium), formado por Affymetryx y Molecular-Pharmacia y a la que se unió posteriormente la empresa Pangea. Su objetivo está en alcanzar una plataforma tecnológica unificada en lo referente a las especificaciones del software. Entre las normativas que deben cumplir se encuentra la estructura de los archivos, el formato TIFF para las imágenes que se extraen de los escáneres, proponiendo e incluso describiendo una arquitectura y diseño de base de datos para estudios de expresión génica.
– Segunda iniciativa a finales de 1998, desde la Life Sciences Research Domain Task Force del Object Management Group. Sus objetivos están centrados en la mejora de la calidad y utilidad del software y sistemas de información para impulsar el desarrollo de herramientas y servicios operativos entre sí en las ciencias de la vida. Otro de sus planteamientos es el de establecer unos estándares para los estudios de la expresión génica.
– Tercera propuesta, surge desde una organización dependiente del gobierno de EE.UU., el NHGRI (Nacional Human Genoma Research Project) y denominada como 15K. Esta estandarización se centra en la definición y establecimiento de una colección física de clones de cDNA, para ser empleados en el diseño y fabricación de los biochips. Este proyecto, por su extensión, se encuentra bajo continua revisión, expansión y desarrollo.
Bibliografía
Petra Dux; Klaas, J. Hellingwerf. Materiales inteligentes para la electrónica del próximo milenio. Universidad de Amsterdam.
Internet “biotic” (Área de Bioinformática y Salud Pública, Instituto de Salud Carlos III).
Microarrays y Biochips de ADN (Genoma España, salud humana).
Wilson, D.S; Nock, S. Functional Protein Microarrays. Current Opinión in Chemical Biology, 6:81 85. 2001.
Nakanishi, T., Oka, T., Akagi, T. Recent advances in DNA microarrays. Acta Med. Okayama, vol. 55, n.º 6:319-328. 2001.
Guillermo H. López Campos, Nieves Ibarrola, Fernándo Martín-Sánchez. Informática y Salud. N.º 25, marzo/abril. 2000.